Zur Erforschung grundlegender Zusammenhänge verwenden wir Systeme mit wohldefinierter Zusammensetzung. Die Mehrzahl unserer Studien konzentriert sich auf die globulären Proteine Myoglobin und Lysozym sowie die fasrigen Proteine Elastin und Kollagen. Zum Beispiel untersuchen wir Elastin mit niedrigem Hydratationsgehalt, um das Crowding in Zellen zu imitieren und die Kristallisation von Wasser in temperaturabhängigen Messungen zu vermeiden. Mittels Neutronenstreuung beobachten wir, dass das mittlere Verschiebungsquadrat der Elastinprotonen dynamische Übergänge bei 3 charakteristischen Temperaturen zeigt. Ein Vergleich mit Ergebnissen aus kernmagnetischer Resonanz und dielektrischer Spektroskopie erlaubt die Folgerung, dass die Übergänge bei 125 K und 195 K durch ein Einsetzen von Methylgruppenreorientierung bzw. Proteinrückgratfluktuationen hervorgerufen werden, die unabhängig von bzw. gekoppelt an die Wasserdynamik erfolgen. Dahingegen lässt sich der Übergang bei 320 K auf den kalorimetrisch beobachteten Glasübergang zurückführen.
Institut für Physik Kondensierter Materie

Dynamik von Proteinen
Für die biologische Funktion von Proteinen ist das Vorhandensein einer Hydrathülle – oder einer geeigneten anderen Lösungsmittelhülle – erforderlich. Das Zusammenspiel von Protein- und Lösungsmitteldynamik bei der biologischen Funktion ist jedoch nur teilweise verstanden.
Makromolekulares Gedränge
Proteine sind in biologischen Zellen gemeinsam mit anderen Makromolekülen dicht gepackt und werden nur von dünnen Lösungsmittelschichten umgeben, die neben Wasser verschiedene weitere Kosolventien enthalten. Aufgrund dieser Komplexität untersuchen wir die Zusammenhänge anhand geeigneter Modellsysteme. Beispielsweise verändern wir für das Bindegewebsprotein Elastin gezielt die Menge und die Zusammensetzung des Lösungsmittels oder imitieren das „Crowding“ in biologischen Zellen durch Einschluss in Poren. In temperaturabhängigen Untersuchungen interessieren wir uns auch für die Eignung verschiedener Kosolventien für die Kryokonservierung von Proteinen.
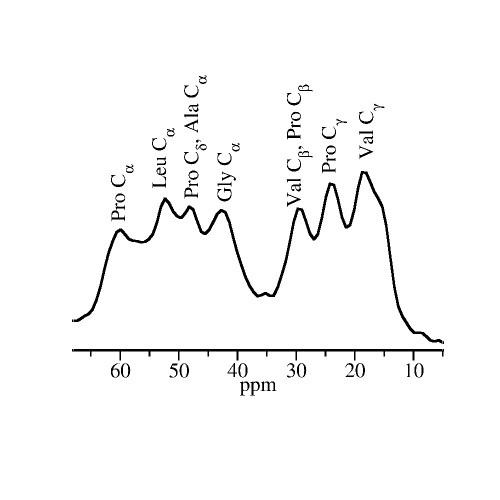
Experimente zur Proteindynamik
In experimentellen Untersuchungen zur Kopplung von Protein- und Lösungsmitteldynamik kombinieren wir kernmagnetische Resonanz, dielektrische Spektroskopie, quasielastische Neutronenstreuung und kalorimetrische Messungen, um ein umfassendes Bild zu gewinnen.
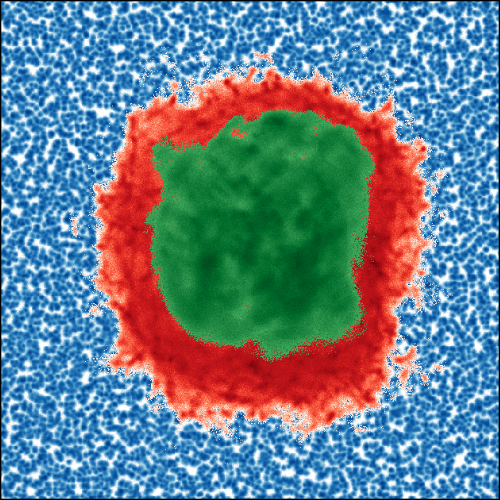
Simulationen zur Proteindynamik
In Molekulardynamik-Simulationen lassen sich die Wechselwirkungen und Zusammensetzungen von Modellsystemen perfekt kontrollieren. Außerdem ist es möglich eine Vielzahl von Eigenschaften des Modells als Funktion von Ort und Zeit zu analysieren. Wir nutzen diese Möglichkeiten zur Erforschung des Zusammenspiels von Protein- und Lösungsmitteldynamik.
Publikationen
- Sorin A. Lusceac, Michael R. Vogel, and Claudia R. Herbers (2010). “2H and 13C NMR Studies on the Temperature-Dependent Water and Protein Dynamics in Hydrated Elastin, Myoglobin and Collagen,” Biochimica et Biophysica Acta (BBA) – Proteins and Proteomics 1804, no. 1 (January 2010): 41–48, https://doi.org/10.1016/j.bbapap.2009.06.009.
- Kerstin Kämpf et al. (2020). “Quasielastic Neutron Scattering Studies on Couplings of Protein and Water Dynamics in Hydrated Elastin,” The Journal of Chemical Physics 152, no. 24 (June 28, 2020): 245101, https://doi.org/10.1063/5.0011107.
- Timothy Wohlfromm and Michael Vogel (2019). “On the Coupling of Protein and Water Dynamics in Confinement: Spatially Resolved Molecular Dynamics Simulation Studies,” The Journal of Chemical Physics 150, no. 24 (June 28, 2019): 245101, https://doi.org/10.1063/1.5097777




